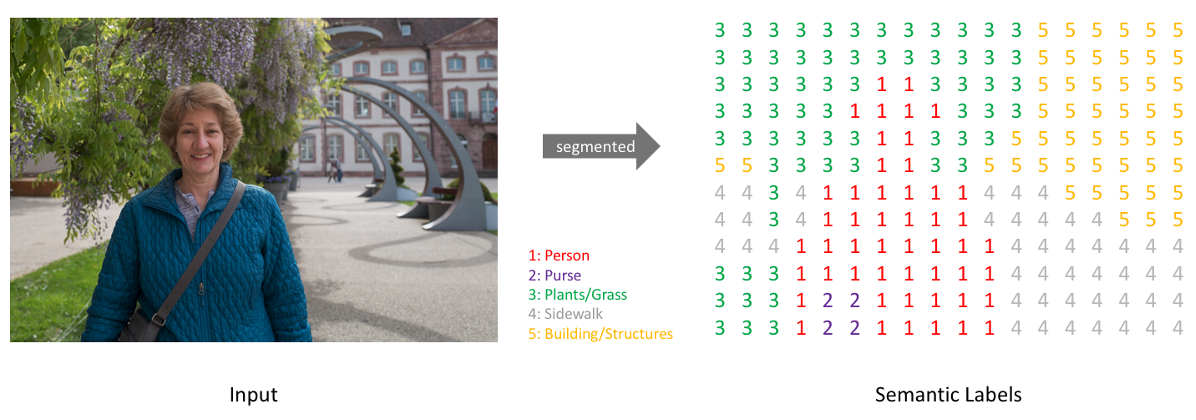
Semantic Segmentation
- 픽셀 기반으로 이미지를 분할하여 구분
0.Short Summary
- 넓은 범위의 이미지 픽셀로부터 의미정보를 추출하고 의미정보를 기반으로 각 픽셀마다 객체를 분류하는 U 모양의 아키텍처
- 서로 근접한 객체 경계를 잘 구분하도록 학습하기 위한 Weighted Loss
1.Introduction
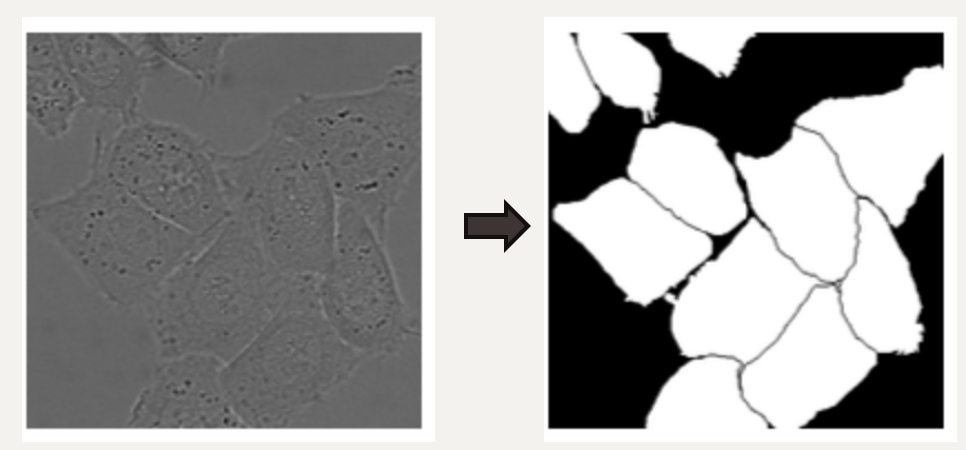
- 생물학 분야의 영상 처리에선 Localization이 포함된 Classification이 필요
- FCN (fully-convolution layer)
- Context(의미정보)를 얻기 위한 Contractinig Path
- Localization(각 픽셀이 어떤 객체에 속하는지)을 위해 Resolution을 키우는 Expanding Path
= 점진적으로 넓은 범위의 이미지 픽셀을 보며 의미정보(Context Information)을 추출하는 수축 경로(Contracting Path)
의미정보를 픽셀 위치정보와 결합(Localization)하여 각 픽셀마다 어떤 객체에 속하는지를 구분하는 확장 경로(Expanding Path)
수축 경로에서 확장 경로로 전환되는 전환 구간(Bottle Neck)
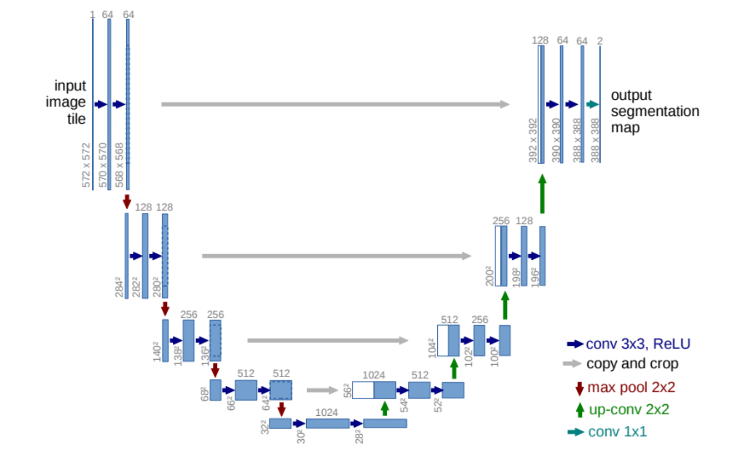
2.Architecture
- 1. Contracting Path (수축경로)

- 논문에서는 BN을 사용하지 않았지만, 대다수의 리뷰 또는 Github에서 BN을 적용한 것을 확인
= 수축경로는 주변 픽셀들을 참조하는 범위를 넓혀가며 이미지로부터 Contextual 정보를 추출하는 역할을 합니다.
3×3 Convolution을 수행할 때 패딩을 하지 않으므로 Feature Map의 크기가 감소합니다.
Downsampling 할 때 마다 채널(Channel)의 수를 2배 증가시키면서 진행합니다.
즉 처음 Input Channel(1)을 64개로 증가시키는 부분을 제외하면 채널은 1−>64−>128−>256−>512−>1024 개로 Downsampling 진행할 때마다 2배씩 증가합니다.
- 2. Bottle Neck (전환 구간)

- 수축 경로에서 확장 경로로 전환되는 구간, Drop out을 통해 모델을 일반화하고, 노이즈에 강하게 해줌
- 3. Expanding Path (확장 경로)

Expanding Path 2)Skip Connection을 통해 수축 경로에서 생성된 Contextual 정보와 위치정보 결합하는 역할을 합니다.
동일한 Level에서 수축경로의 Feature Map과 확장경로의 Feature Map의 크기가 다른 이유는 여러번의 패딩이 없는 3×3 Convolution Layer를 지나면서 특징맵의 크기가 줄어들기 때문입니다.
Expanding Path의 마지막에 Class의 갯수만큼 필터를 갖고 있는 1×1 Convolution Layer가 있습니다.
1×1 Convolution Layer를 통과한 후 각 픽셀이 어떤 Class에 해당하는지에 대한 정보를 나타내는 3차원(Width × Height × Class) 벡터가 생성됩니다.
3.Training
- 1. Overlap-tile strategy
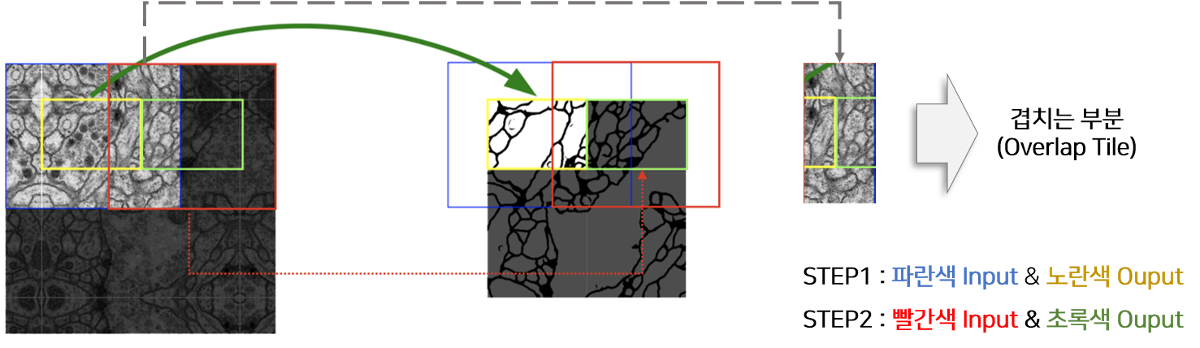
본 논문에서 다양한 학습 장치들을 통해 모델의 성능을 향상시킵니다.
이미지의 크기가 큰 경우 이미지를 자른 후 각 이미지에 해당하는 Segmentation을 진행해야 합니다.
U-Net은 Input과 Output의 이미지 크기가 다르기 때문에 위 그림에서처럼 파란색 영역을 Input으로 넣으면 노란색 영역이 Output으로 추출됩니다
동일하게 초록색 영역을 Segmentation하기 위해서는 빨간색 영역을 모델의 Input으로 사용해야 합니다.
즉, 겹치는 부분이 존재하도록 이미지를 자르고 Segmentation하기 때문에 Overlap Tile 전략이라고 논문에서는 지칭합니다.
= Fully Convolutional Network 구조의 특성상 입력 이미지의 크기에 제약이 없다. 따라서 U-Net 연구팀은 크기가 큰 이미지의 경우 이미지 전체를 사용하는 대신 overlap-tite 전략을 사용
- 2. Mirroring Extrapolate
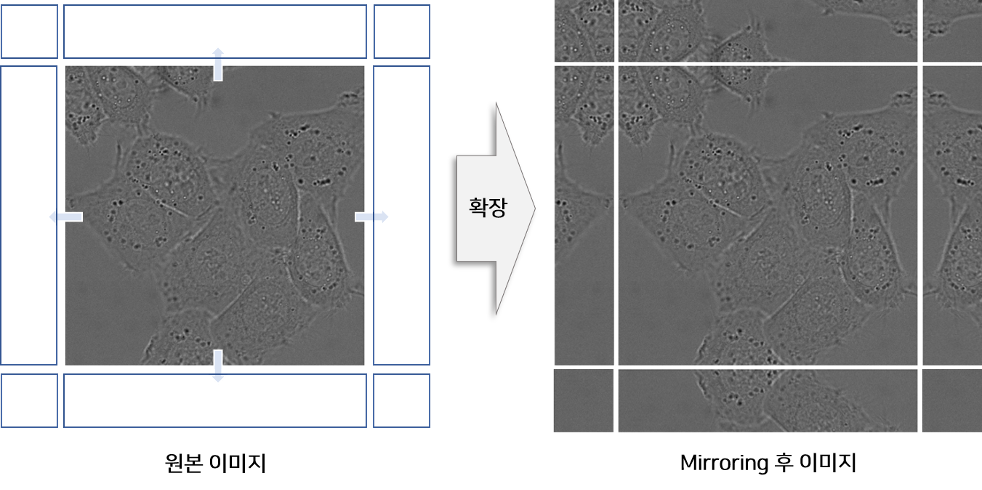
이미지의 경계부분을 예측할 때에는 Padding을 넣어 활용하는 경우가 일반적입니다. 본 논문에서는 이미지 경계에 위치한 이미지를 복사하고 좌우 반전을 통해 Mirror 이미지를 생성한 후 원본 이미지의 주변에 붙여 Input으로 사용합니다.본 논문의 실험분야인 biomedical 에서는 세포가 주로 등장하고, 세포는 상하 좌우 대칭구도를 이루는 경우가 많기 때문에 Mirroring 전략을 사용했을 것이라고 추측합니다.
- 3. Weighted Loss
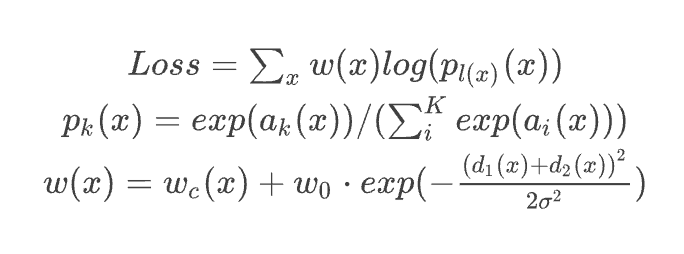
- x는 feature map에 있는 각 픽셀
- w(x)는 weight map, 픽셀 별로 가중치를 부과하는 역할

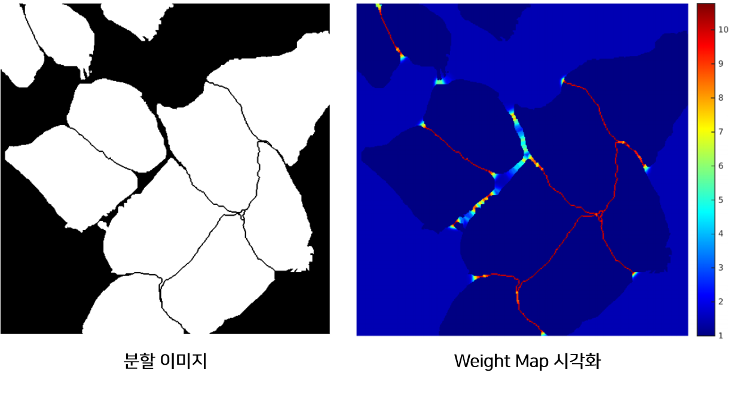
모델은 위 그림처럼 작은 경계를 분리할 수 있도록 학습되어야 합니다. 따라서 논문에서는 각 픽셀이 경계와 얼마나 가까운지에 따른 Weight-Map을 만들고 학습할 때 경계에 가까운 픽셀의 Loss를 Weight-Map에 비례하게 증가 시킴으로써 경계를 잘 학습하도록 설계하였습니다.
Loss는 맨 마지막에 얻은 feature map에 픽셀 단위로 softmax를 수행하고 여기에 cross entropy loss function을 적용하는 식
weight map을 의미하는 w(x)는 특정 클래스가 가지는 픽셀의 주파수의 차이를 보완해주는 식이라고 보시면 됩니다. 같은 세포 클래스라도 픽셀값에 차이가 있으니 그런걸 채워준다 뭐 그런뜻이 아닌가 싶습니다.
weight map에 픽셀에서 얻은 클래스별 예측값을 soft-max한 것의 log를 곱한 것이 E라고 할 수 있겠습니다.
- 4. Data Augmentation
- shift, rotation, random-elastic deformation 기법을 사용
- elastic deformation ?
“We generate smooth deformations using random displacement vectors on a coarse 3 by 3 grid. The displacements are sampled from a Gaussian distribution with 10 pixels standard deviation. Per-pixel displacements are then computed using bicubic interpolation. Drop-out layers at the end of the contracting path perform further implicit data augmentation.”
4.Experiment
1. EM Segmentation challenge
EM Segmentation challenge의 Dataset을 활용, 약 30개의 data (이미지 + GT)
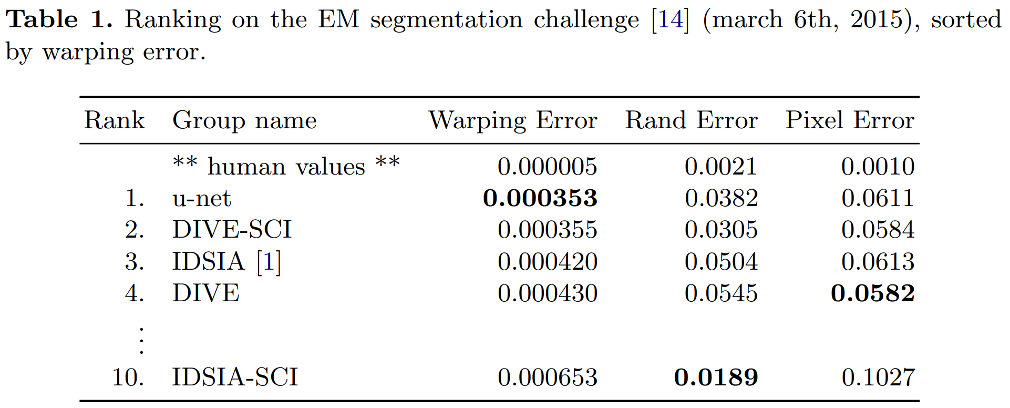
- Warping Error : 객체 분할 및 병합이 잘 되었는지를 확인하는 segmentation과 관련된 에러
2. EM Segmentation challenge
- Iou score 사용, 세포라고 구분한 영역과 실제 세포의 영역이 겹치는 정도로 성능을 측정
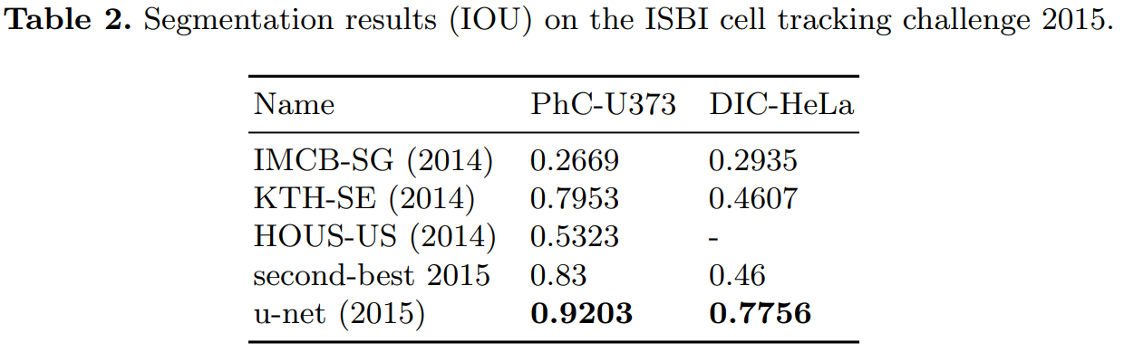
세포 분류 대회인 ISBI cell tracking challeng 에서 모델의 성능을 평가한 표는 아래와 같습니다.
“PhC-U373” 데이터는 위상차 현미경으로 기록한 35개의 이미지를 Training 데이터로 제공합니다.
“DIC-HeLa” 데이터는 HeLa 세포를 현미경을 통해 기록하고 20개의 이미지를 Training 데이터로 제공합니다.
5.Conclusion
U-Net은 다양한 biomedical segmentation applications에서 "아주" 좋은 성능을 보여줬습니다.
저자는 elastic deformation이 포함된 Data augmentation 덕분에 적은 사이즈의 데이터셋만 요구했고 합리적인 학습 시간(NVidia Titan GPU (6 GB)에서 10시간 학습)을 가졌다고 말했습니다.
그리고 마지막으로 U-Net의 구조가 다양한 task에 쉽게 응용될 수 있을거라 확신한다고 말하며 논문을 끝냅니다.
Skip Architecture는 Layer를 깊게 쌓을수 있게 하여 복잡한 Task를 잘 수행할 수 있게 합니다.
또한 Bottle Neck에서 생기는 정보의 손실을 줄이는 역할을 합니다.
Weighted Loss는 근거리에 있는 객체를 효과적으로 분리하여 학습하는 좋은 방법